植物における免疫誘導と病原微生物の感染戦略
2013/08/13
川崎 努
(近畿大学農学部 バイオサイエンス学科植物分子遺伝学研究室)
email:川崎 努
領域融合レビュー, 2, e008 (2013) DOI: 10.7875/leading.author.2.e008
Tsutomu Kawasaki: Plant immunity and inhibitory mechanism of host immunity by pathogens.

植物は細胞膜に存在する病原微生物の認識にかかわる受容体により,侵入してきた病原微生物の構成成分を検出し,さまざまな免疫応答を誘導する.近年,病原微生物の認識にかかわる受容体の構造と機能が明らかにされるとともに,それら受容体と複合体を形成するタンパク質が同定され,病原微生物の認識にともなう免疫誘導の分子機構が明らかになってきた.一方,病原微生物は植物の免疫誘導を抑制するため,エフェクターとよばれる自らのタンパク質を植物の細胞内に分泌し,病原微生物の認識にかかわる受容体を介した免疫反応の誘導を阻害していることがわかってきた.ここでは,受容体を介した植物の免疫誘導の分子機構と,エフェクターを利用した病原微生物の感染戦略に関して,最新の知見を解説する.
毎年,世界の作物生産の約15%は病害により失われている.一方で,世界的に爆発的な人口の増加が予測されており,国連食糧農業機関の発表によると,2050年には世界の人口は90億人をこえ,現在より食糧の60%の増産が必要であると試算されている.しかし,単位面積あたりの作物の生産量を大幅に向上させることは非常にむずかしく,病害による15%の損失をいかに抑えるかが今後の重要な課題のひとつになっている.
野菜や庭木などを育てていると,いろいろな病気が発生しその対策に悩まされる.植物を侵している病原微生物も,細菌,カビ,ウイルスなど,さまざまである.病気になった植物をみていると,あたかも植物が病原微生物から一方的な攻撃をうけているようにみえるが,本当にそうであろうか.地球上には多くの病原微生物が存在しているが,それらがすべての植物に感染できるわけではなく,個々の病原微生物は特定の植物にしか感染できない.つまり,植物は多くの病原微生物の感染を阻止しているのである.では,植物はどのようにして病原微生物の感染を阻止しているのだろうか.動物では一般に“免疫”として,病原微生物の感染にともない防御応答を誘導することが知られている.植物も動物の免疫と同じような方法で病原微生物に対し防御応答を誘導しているのだろうか.
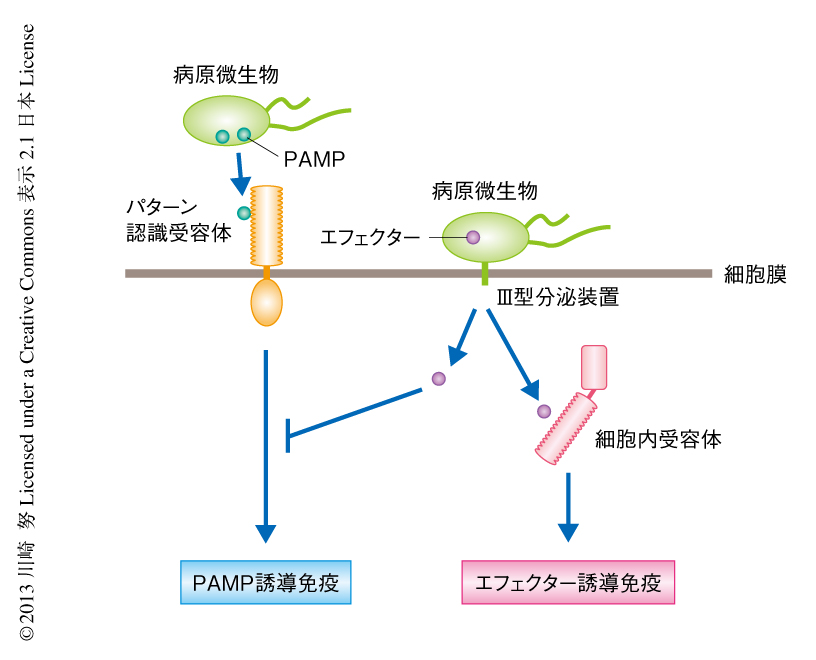
動物の免疫応答は自然免疫と獲得免疫とに大別される.獲得免疫は古くからよく知られている抗原抗体反応にもとづく免疫反応であり,病原微生物の感染から抗体ができるまで数週間を要する.一方,自然免疫は病原微生物の感染にともない迅速に誘導される免疫応答である.自然免疫反応では,宿主(感染をうける側を宿主という)の細胞膜に存在する受容体が,病原微生物の構成成分(PAMP:pathogen-associated molecular pattern,病原微生物関連分子パターン)を検出することにより誘導される1)(図1).代表的なPAMPとして細菌のべん毛タンパク質やリポ多糖などが知られている.動物の細胞の表面にはこのようなPAMPを認識するセンサー(受容体)が存在し,それらは総称してパターン認識受容体(pattern recognition receptor:PRR)とよばれている.また,パターン認識受容体がPAMPを検出することにより誘導される免疫反応をPAMP誘導免疫(PAMP-triggered immunity:PTI)とよぶ.動物のパターン認識受容体の代表としてToll様受容体(Toll-like receptor:TLR)が知られており,Bruce A. BeutlerとJules A. HoffmannはToll様受容体の発見により2011年にノーベル生理学・医学賞を受賞している.ヒトには11個のToll様受容体が存在し,それらがさまざまな種類のPAMPを認識しそのシグナルを細胞内に伝達することにより,病原微生物の感染に対し迅速な免疫反応の誘導を可能にしている.
一方,植物には動物の獲得免疫のような抗原抗体反応にもとづく防御応答は存在しない.しかし,植物も動物と同様にパターン認識受容体をもち,PAMPを検出して防御応答を誘導することができる.その反応は動物の自然免疫に非常に似ていることから,近年,“植物免疫”とよばれている.わが国ではまだこの植物免疫という言葉にはなじみがないように思われるが,国際的には“plant immunity”という用語はすでに定着している.
病原微生物にとり,感染した細胞において自らのPAMPが宿主のパターン認識受容体に検出されてしまうと防御応答をうけることになり,増殖することができなくなる.しかし,病原微生物はただ手をこまねいているわけではない.病原微生物は進化により宿主の免疫応答を阻止することのできるタンパク質(エフェクターとよばれる)を獲得し,それを宿主の細胞内に送り込むことにより防御応答からまぬがれている1)(図1).それに対しさらに植物は,病原微生物が送りこんできたエフェクターを検知する細胞内受容体を獲得し,PAMP誘導免疫と比較してより強い免疫反応を誘導する.この反応はエフェクター誘導免疫(effector-triggered immunity:ETI)とよばれている(図1).では,このような植物の免疫反応と病原微生物による免疫反応の阻害はどのように行われているのだろうか.くわしくみてみよう.
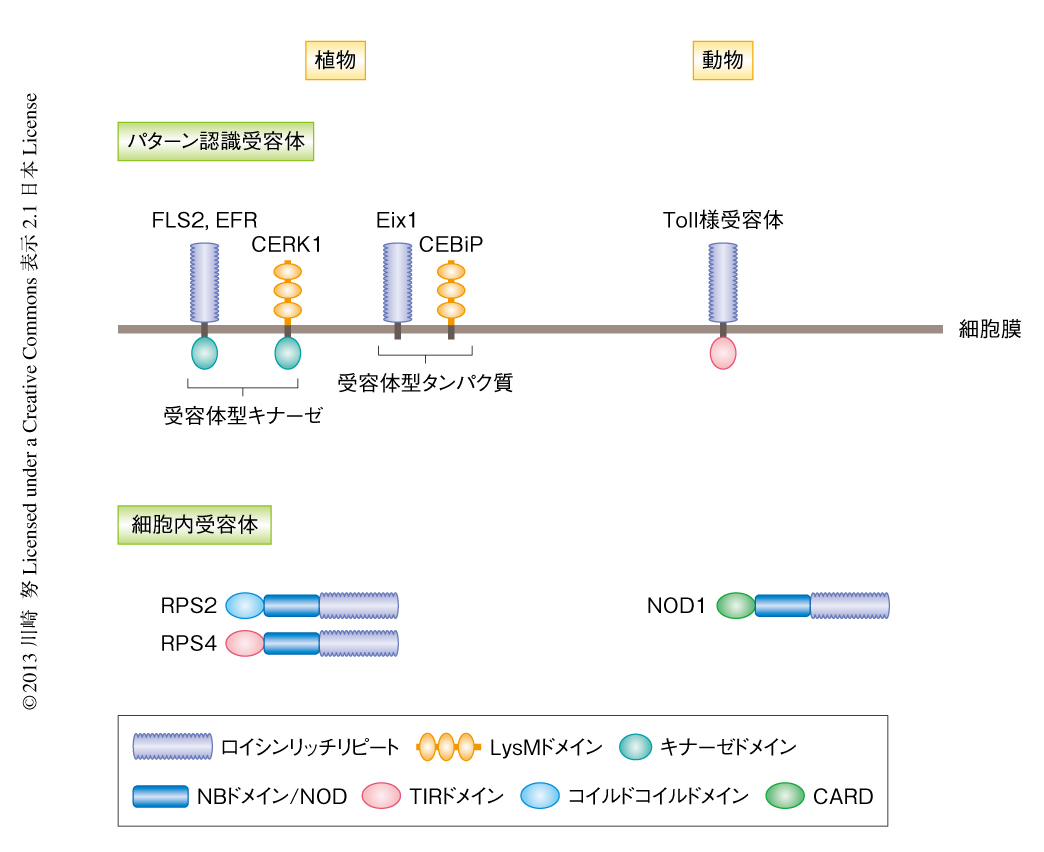
植物の細胞膜に存在するパターン認識受容体は,受容体型キナーゼ(receptor-like kinase:RLK)と受容体型タンパク質(receptor-like protein:RLP)とに大きく分類される2)(図2).受容体型キナーゼはPAMPを検出する細胞外ドメインと細胞内にタンパク質キナーゼドメインをもつ受容体であり,一方,受容体型タンパク質は細胞外ドメインをもつものの細胞内の領域は短く特徴的なドメインをもたない.2000年以降,植物では細菌のべん毛タンパク質に由来するペプチド(flg22),翻訳伸長因子に由来するペプチド(elf18),細胞壁成分であるペプチドグリカン,そして,真菌のもつキチンを検出するパターン認識受容体が明らかになり先行して研究が進められているが,現在でも,パターン認識受容体とPAMPとの組合せが明らかになっているものはそれほど多くない.パターン認識受容体のもつ代表的な細胞外ドメインとしては,ロイシン残基のくり返し配列であるロイシンリッチリピート(leucine rich repeat:LRR),キチンやペプチドグリカンなどのオリゴ糖と結合するLysMドメインなどがある.植物のパターン認識受容体とヒトのToll様受容体の構造をみてみると,Toll様受容体のもつ細胞外ドメインはロイシンリッチリピートであり,植物のパターン認識受容体と非常によく似ていることがわかる(図2).
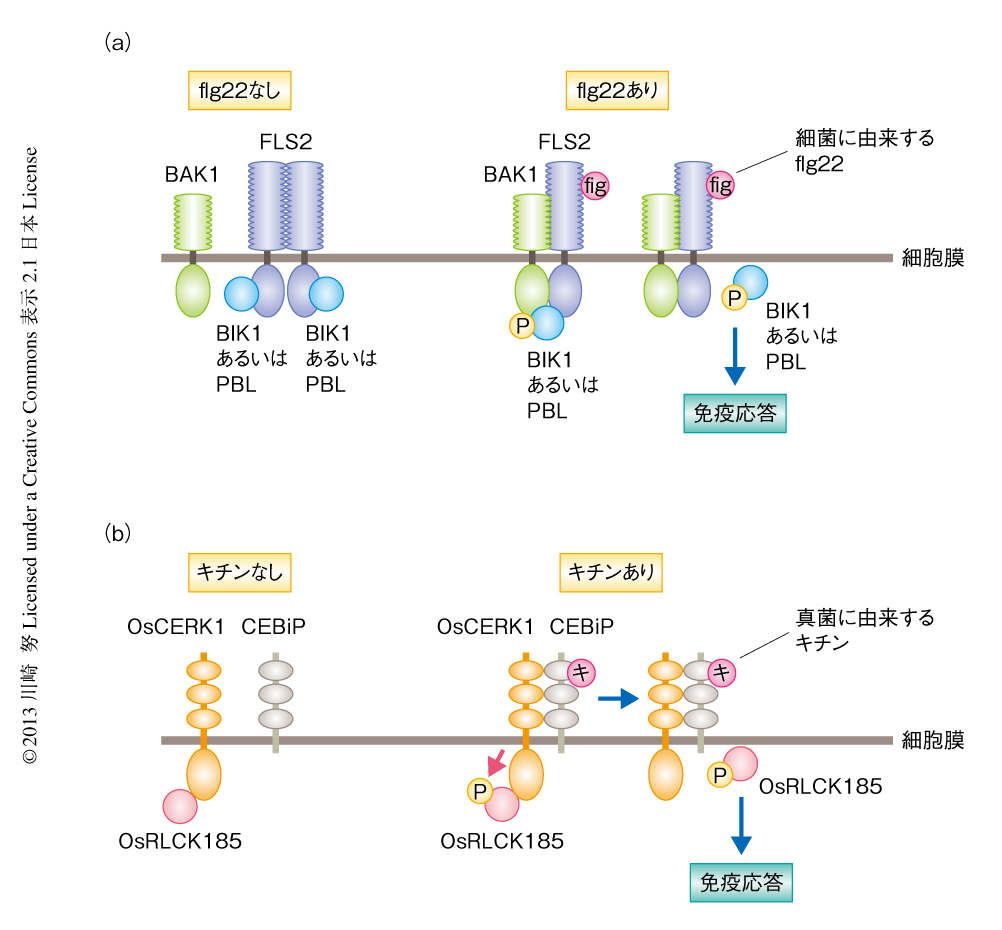
つぎに,このようなパターン認識受容体がどのようにPAMPを検出し,そのシグナルを細胞内に伝達するかをみてみよう.植物でもっともよく解析されているパターン認識受容体は,モデル植物であるシロイヌナズナ(Arabidopsis thaliana)のFLS2である.FLS2は細胞外にロイシンリッチリピートを,細胞内にタンパク質キナーゼドメインをもち,細菌のべん毛タンパク質のN末端領域に高度に保存されている22個のアミノ酸残基からなるペプチドflg22を検出する(図3).FLS2はflg22の非存在下では細胞膜においてホモ複合体を形成しているが3),flg22を検出するとやはり細胞外にロイシンリッチリピートをもつ受容体型キナーゼであるBAK1と相互作用する4).このFLS2とBAK1との相互作用は,細胞にflg22を処理したのち数分以内に起こることが報告されている.この相互作用によりFLS2とBAK1は相互にリン酸化され活性化状態になると考えられる.そののち,FLS2からのシグナルは受容体型細胞質タンパク質キナーゼ(RLCK:receptor-like cytoplasmic kinase)ファミリーに属するBIK1,PBS1,PBL1,PBL2,BSK1に伝達される5,6).これらRLCKファミリータンパク質はflg22の処理にともないリン酸化されることが明らかになっているが,FLS2とBAK1のどちらがRLCKをリン酸化しているかについては議論があり最終的な結論は得られていない.いずれにしても,これらのRLCKファミリータンパク質が受容体からのシグナルを受け取り,そのシグナルを細胞内に伝達していることはまちがいないと考えられる.ほかの植物にもFLS2やBAK1のホモログは存在するが,同様な機構によりflg22を認識しているのかどうかはわかっていない.また,ヒトのToll様受容体であるTLR5はべん毛タンパク質を検出するが,flg22とは異なるべん毛タンパク質の領域を検出することが明らかになっている.
真菌の成分であるキチンは代表的なPAMPとして植物に強い免疫応答を誘導する.イネにおけるキチンの検出にはパターン認識受容体であるOsCERK1とCEBiPが関与している(図3).OsCERK1は細胞外にLysMドメイン,細胞内にタンパク質キナーゼドメインをもち,CEBiPは細胞外にLysMドメインをもつものの,細胞内の領域は短く特異的なドメインはもたない7).CEBiPはキチンとの強い結合活性をもち,キチンを検出したCEBiPはOsCERK1と相互作用する.このことから,OsCERK1がキチンを検出したCEBiPからシグナルをうけ,そのシグナルを細胞内に伝達していると考えられていたが,その詳細は不明であった.筆者らは,OsCERK1の細胞内のタンパク質キナーゼドメインに結合するタンパク質として,RLCKファミリーに属するOsRLCK185を同定した8)(新着論文レビュー でも掲載).そして実際に,細胞膜においてOsCERK1とOsRLCK185とが相互作用していることを示した.さらに,OsRLCK185の発現をノックダウンしたイネの培養細胞ではキチンに応答した免疫応答が抑制され,OsRLCK185がOsCERK1からのシグナルを伝達する役割をはたしていることが明らかになった.
OsRLCK185はOsCERK1のタンパク質キナーゼドメインと結合していたことから,OsCERK1からOsRLCK185へのシグナル伝達はOsCERK1がOsRLCK185をリン酸化することにより行われている可能性が考えられた.そこで,キチンに応答したOsRLCK185のリン酸化について調べてみると,イネの培養細胞にキチンを処理したのち約5分でOsRLCK185はリン酸化されていた.さらに,OsCERK1およびOsRLCK185の組換えタンパク質を用いた解析により,OsCERK1がOsRLCK185を直接にリン酸化することが明らかになった.また一般に,RLCKファミリータンパク質は活性化ループとよばれる領域をもち,その活性化ループに存在するセリン残基とスレオニン残基のリン酸化が活性化に重要であると報告されている.そこで,OsRLCK185のもつ活性化ループのセリン残基とスレオニン残基をアラニン残基に置換したうえで,OsCERK1によるリン酸化実験を行ったところ,このOsRLCK185変異体はリン酸化されなかった.すなわち,OsCERK1はOsRLCK185の活性化ループに存在するセリン残基とスレオニン残基をリン酸化することによりOsRLCK185を活性化していると考えられた.
このように,キチンにより誘導される免疫応答では,キチンを受容したCEBiPがOsCERK1にシグナルを伝達し,OsCERK1はOsRLCK185を直接にリン酸化することにより細胞内にシグナルを伝達していることが明らかになった.このように,FLS2やOsCERK1などのパターン認識受容体からのシグナルはRLCKファミリータンパク質に伝達されていることから,ほかのパターン認識受容体においても同様にRLCKファミリータンパク質を介して細胞内にシグナルが伝達されている可能性が考えられる.イネには379個のRLCKファミリー遺伝子が存在し9),シロイヌナズナにも200個のRLCKファミリー遺伝子が存在しているが10),ほとんどのRLCKファミリータンパク質の機能は明らかになっておらず,今後の研究が期待される.
最近,OsCERK1から細胞内シグナル伝達には,低分子量Gタンパク質であるOsRac1を介したシグナル伝達も寄与していることが明らかになった11)(新着論文レビュー でも掲載).これまでの研究により,OsRac1はさまざまな免疫応答を誘導するシグナル伝達タンパク質と機能していることが示されており,OsCERK1は,OsRac1を活性化するGDP-GTP交換因子(GDP-GTP exchange factor:GEF)を直接にリン酸化することにより,OsRac1を介したシグナル伝達を活性化していることが明らかになった.
シロイヌナズナではOsCERK1のホモログであるAtCERK1がキチンを認識することが明らかになっている.イネのOsCERK1のもつLysMドメインはキチンとは結合しないためキチンに結合したCEBiPからシグナルを受け取るが,シロイヌナズナのAtCERK1のもつLysMドメインはキチンと強く結合し,ホモ複合体を形成することによりキチンを認識していることが明らかになった12).また,CEBiPと似た構造をもつシロイヌナズナのAtLYM1とAtLYM3は,PAMPのひとつである細菌の細胞壁成分ペプチドグリカンと結合し,そのシグナルをAtCERK1を介し細胞内に伝達していることが明らかになった13).イネにおいても,AtLYM1およびAtLYM3のホモログであるOsLYP4およびOsLYP6がペプチドグリカンと結合することが明らかになっており14),シロイヌナズナと同様に,OsLYP4とOsLYP6がペプチドグリカンを検出し,そのシグナルをOsCERK1を介して細胞内に伝達していると推測されているが,実際に,OsCERK1がペプチドグリカンの認識にかかわるかどうかは証明されていない.しかし,OsRLCK185のノックダウン株ではペプチドグリカンに応答した免疫応答が抑制されており,OsCERK1がペプチドグリカンの認識に関与することが強く示唆されている.このように,CERK1は真菌のPAMPであるキチンおよび細菌のPAMPであるペプチドグリカンを検出するパターン認識受容体であり,真菌と細菌の両方に対し免疫応答を誘導していると考えられる.
筆者らは,OsRLCK185のシロイヌナズナにおけるオーソログとしてAtRLCK1を同定している.AtRLCK1は,イネのOsRLCK185と同様にCERK1に結合し,AtRLCK1の機能欠損変異体ではキチンに応答した免疫応答は阻害されていることがわかった(未発表).このように,イネとシロイヌナズナにはキチンに対する応答に関与する共通のシグナル伝達系が存在していると思われる.
植物はパターン認識受容体により病原微生物の感染を認識し,さまざまな免疫応答を誘導して病原微生物の増殖を阻止している.では,病原微生物はこの植物の免疫応答にどのように対抗しているのだろうか.病原微生物は宿主への感染過程において自らのタンパク質を宿主の細胞内に分泌し免疫応答を抑制している(図1).このような分泌タンパク質は総称してエフェクターとよばれている.病原微生物の種類によりエフェクターの分泌の様式は異なるが,ここでは,一部の病原細菌が利用するIII型分泌装置について紹介したい.Pseudomonas属やXanthomonas属の病原細菌は針状の構造をしたIII型分泌装置を用いて宿主の細胞内にエフェクターを分泌している.III型分泌装置を構成するタンパク質をコードする遺伝子はhrp(hypersensitive response and pathogenicity)遺伝子とよばれ,このhrp遺伝子は植物へ感染するときのみ発現する.つまり,病原細菌は宿主の組織内の環境を感知してIII型分泌装置を構築し,宿主の細胞内にエフェクターを送り込んでいるのである.
Pseudomonas属やXanthomonas属の細菌は30~50個のエフェクターをもち,III型分泌装置を使いこれを宿主の細胞内に送り込んでいる.しかし,アミノ酸配列から機能を推定できるエフェクターは少なく,作用機作のわかっているエフェクターの数も少ない.また,エフェクターの多くは既知の機能ドメインをもたないことから,進化的に新しいタンパク質であると考えられている.実際,近年になり,エフェクターの研究から新規な酵素活性が見い出されている.また,多くのエフェクターは宿主の細胞内で発現させるとその免疫応答を強く阻害する.このことは,宿主において免疫応答の誘導に重要な因子の機能をエフェクターが阻害していることを示唆している.そのため近年,エフェクターが標的とする因子を探索することにより,免疫応答において重要な役割をはたしている因子を同定しようという試みが行われている.その具体例について紹介していこう.
斑葉細菌病をひき起こすPseudomonas syringaeはグラム陰性細菌であり,古くからシロイヌナズナを用いた免疫の研究において代表的な病原微生物として利用されている.P. syringaeのエフェクターHopM1の標的となる因子として,シロイヌナズナのMIN7遺伝子がクローニングされた15).MIN7遺伝子はARFのGDP-GTP交換因子をコードしており,PAMP誘導性免疫やエフェクター誘導免疫に関与する小胞輸送を制御することが示唆されている16).また,P. syringaeのエフェクターHopU1の標的となる因子として,シロイヌナズナにおいてRNA結合タンパク質をコードするGRP7遺伝子がクローニングされた17).HopU1はGRP7のRNA認識モチーフに存在するアルギニン残基をADPリボース化することにより,GRP7のRNA結合活性を阻害していることが報告された18).さらに最近,GRP7はパターン認識受容体であるFLS2の転写産物や翻訳関連タンパク質と相互作用することにより転写後制御に関与しており19),HopU1はGRP7とFLS2の転写産物とのあいだの相互作用を阻害することにより,FLS2のタンパク質量を減少させていることが報告された.
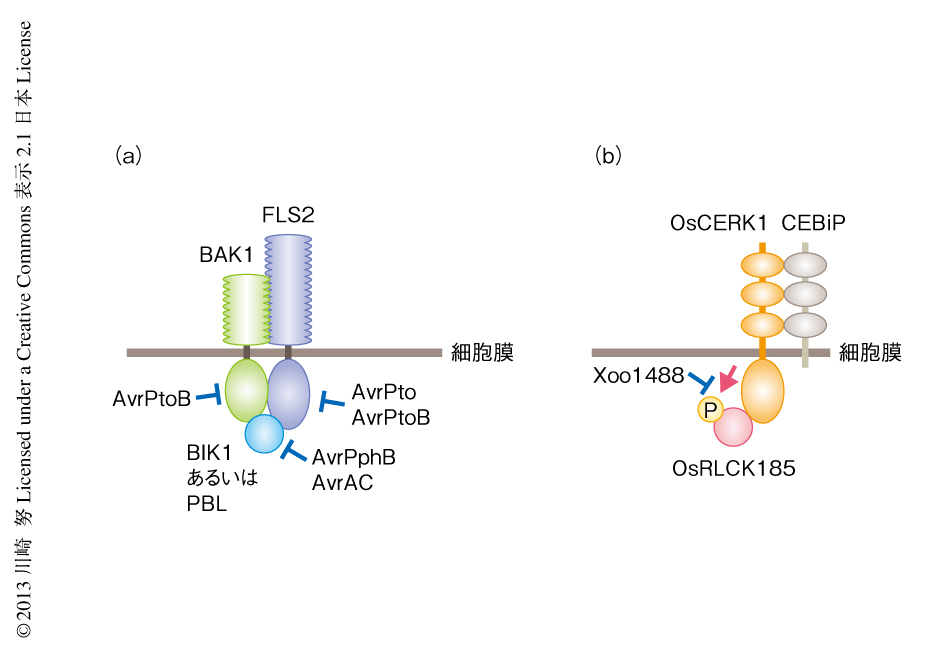
FLS2やCERK1による免疫誘導機構の解析から明らかになったように,RLCKファミリータンパク質はパターン認識受容体からのシグナルを細胞内に伝達する重要なタンパク質である.近年,エフェクターが相互作用するタンパク質の探索により,RLCKファミリータンパク質がさまざまなエフェクターの標的となることも明らかになってきた.P. syringaeのエフェクターAvrPphBはシステインプロテアーゼ活性をもち,免疫応答に関与するRLCKファミリータンパク質であるBIK1やPBS1,PBL1,PBL2を切断することにより免疫応答を抑制していることが明らかになった5)(図4).また,Xanthomonas campestrisのエフェクターであるAvrACは,RLCKファミリーに属するRIPKおよびBIK1を標的としていることが明らかになった20).RLCKファミリータンパク質の活性化においてその活性化ループに存在するセリン残基やスレオニン残基のリン酸化が重要であると考えられるが,AvrACはそのリン酸化部位にウリジル酸を付加することによりRIPKおよびBIK1の活性化を阻害していた.また,RLCKファミリーのほかのタンパク質においても活性化ループのリン酸化部位は高度に保存されており,それらがAvrACの標的となる可能性も考えられる.また,筆者らは,イネ白葉枯病菌Xanthomonas oryzaeのエフェクターであるXoo1488がOsRLCK185を標的としていることを明らかにした.OsCERK1はOsRLCK185をリン酸化することによりシグナルを伝達しているが,Xoo1488はOsCERK1によるOsRLCK185のリン酸化を阻害していた8)(新着論文レビュー でも掲載).このように,複数のエフェクターがRLCKファミリータンパク質を標的としていることが明らかになり,このことは,RLCKファミリータンパク質が免疫応答において重要な役割をはたしていることを裏づけるものと考えられる.
P. syringaeのエフェクターHopZ1は抗菌性物質であるダイドゼインの合成に関与する2-ヒドロキシイソフラバノンデヒドラターゼを標的としていることが明らかになった21).免疫応答の際に産生される抗菌性物質はファイトアレキシンとよばれ,その産生は免疫応答における重要な反応のひとつである.HopZ1はファイトアレキシンの産生を抑制することにより免疫応答を阻害していると考えられる21).
また,エフェクターがパターン認識受容体を直接に抑制することも報告されている(図4).P. syringaeのエフェクターであるAvrPtoおよびAvrPtoBは,FLS2やEFR,CERK1と直接に相互作用し免疫誘導を阻害していることが示された22-25).さらに,AvrPtoBはBAK1も阻害していることが明らかになった26).BAK1は複数のパターン認識受容体と協調して機能していることから,病原微生物にとりBAK1の機能を抑制することにより複数のパターン認識受容体を阻害することが可能になり,免疫応答を効率的に抑制することができるものと考えられる.
Xanthomonas属の細菌はTAL(transcription activator-like)とよばれるエフェクターをもつ.このTALエフェクターは宿主のもつDNA配列を特異的に認識する反復配列と,核移行シグナルおよび転写活性化ドメインをもつ転写因子である.反復配列は34アミノ酸残基からなり,その12番目と13番目のアミノ酸残基が認識する塩基配列の特異性を決めている.最近,X. oryzaeのTALエフェクターであるAvrXa7は,SWEETSとよばれる糖輸送体Os8N3およびOs11N3の遺伝子の転写を活性化することが報告された27).AvrXa7は自らの増殖に必要な養分を得るため,宿主のもつ糖輸送体を活性化していると考えられる.また,TALエフェクターのDNA結合領域を利用すると,任意の塩基配列に結合するDNA結合タンパク質を構築することができる.これを使い人工酵素であるTALエフェクターヌクレアーゼ(transcription activator-like effector nuclease:TALEN)を作製し,それを細胞に導入することにより目的の遺伝子を改変した生物が作出できるようになった.このように,TALエフェクターは宿主において転写を制御することにより免疫応答の抑制,あるいは,自らの増殖に適した環境をつくりだしている.
以上のように,個々のエフェクターの機能が少しずつ明らかにされ,さらに,エフェクターを分子プローブとした新規な因子の探索により,これまで明らかにされていなかった免疫にかかわる因子が同定されている.しかし,解析されたエフェクターはまだ一部にすぎず,いまだ,多くのエフェクターの機能,あるいは,宿主における標的は不明である.宿主と病原微生物との攻防を分子レベルで解明するためには,さらなる研究が必要である.
植物および動物の細胞膜にはパターン認識受容体が存在しPAMPを検出して防御応答を誘導していることを説明してきたが,植物および動物は細胞内にも共通の構造をもつ細胞内受容体をもつ(図2).この細胞内受容体は中心部に核酸結合ドメイン,C末端側にロイシンリッチリピートをもつ.この核酸結合ドメインは,植物ではNBドメイン(NB:nucleotide-binding),動物ではNOD(nucleotide-binding oligomerization domain,ヌクレオチド結合オリゴマー形成ドメイン)とよばれている1).植物の細胞内受容体の多くはN末端側にコイルドコイルドメインあるいはTIRドメインをもつ.一方,動物の細胞内受容体のN末端側にはカスパーゼ結合ドメイン(caspase recruitment domain:CARD)などが存在する.これらN末端側の領域はタンパク質間相互作用に関係し,下流にシグナルを伝達するためのドメインとして機能していると考えられており,動物ではCARDを介した免疫応答の活性化機構が明らかにされている.しかし,植物では細胞内受容体から下流にどのようにシグナルが伝達されているか,ブラックボックスのままである.
植物の細胞内受容体はもともと,病原微生物の感染にともない強い抵抗性反応を誘導する抵抗性遺伝子座にコードされるタンパク質(抵抗性タンパク質,あるいは,Rタンパク質ともよばれる)として同定された.のちの研究により,抵抗性タンパク質として機能する細胞内受容体は病原微生物のエフェクターを特異的に認識し,過敏感細胞死をともなう強い免疫反応(エフェクター誘導免疫)を誘導することが明らかになった(図1).植物の細胞内受容体の認識においては,細胞内受容体とエフェクターとが直接的に相互作用する場合と,エフェクターと宿主のほかのタンパク質との相互作用を細胞内受容体が検出することによりエフェクターを間接的に認識する場合とがある.たとえば,シロイヌナズナの細胞内受容体であるRPS5はP. syringaeのエフェクターAvrPphBがRLCKファミリータンパク質であるPBS1を切断するのを検出して免疫反応を誘導する28).また,P. syringaeのエフェクターAvrBはRLCKファミリータンパク質であるRIPKによるRIN4のリン酸化を促進し,細胞内受容体であるRPM1はリン酸化されたRIN4を検出することにより免疫応答を誘導している29).このように,植物の細胞内受容体はエフェクターを検出して免疫を誘導しているが,動物の細胞内受容体ではエフェクターではなくPAMPを検出して免疫応答を誘導することが知られている.今後,研究が進むにつれ,植物の細胞内受容体によるPAMPの認識,あるいは,動物の細胞内受容体によるエフェクターの認識が明らかになるかもしれない.
MAPキナーゼは酵母から植物あるいは動物にいたるまで真核生物に広く保存されたセリン/スレオニンキナーゼである.MAPキナーゼは免疫応答を含むさまざまな生理反応において活性化され,活性化されたMAPキナーゼは転写因子や酵素などをリン酸化することにより細胞内シグナル伝達を担うモジュールとして機能している.MAPキナーゼはMAPキナーゼキナーゼ(MAPKK)がキナーゼサブドメインVIIとキナーゼサブドメインVIIIとのあいだにあるTxYモチーフのスレオニン残基およびチロシン残基をリン酸化することにより活性化される.さらに,MAPKKはMAPキナーゼキナーゼキナーゼ(MAPKKK)がキナーゼサブドメインVIIとキナーゼサブドメインVIIIとのあいだにあるSxxxS/Tモチーフのセリン残基およびスレオニン残基をリン酸化することにより活性化される30).これら3種類のタンパク質キナーゼのリン酸化によるシグナル伝達はMAPキナーゼカスケードとよばれている(図5).シロイヌナズナには60個のMAPKKK,10個のMAPKK,20個のMAPキナーゼが,イネには75個のMAPKKK,8個のMAPKK,15個のMAPキナーゼが同定されており,その数の多さから複雑なカスケードが想像できるが,どのようにシグナルが伝達されているかについて,その詳細は不明である.
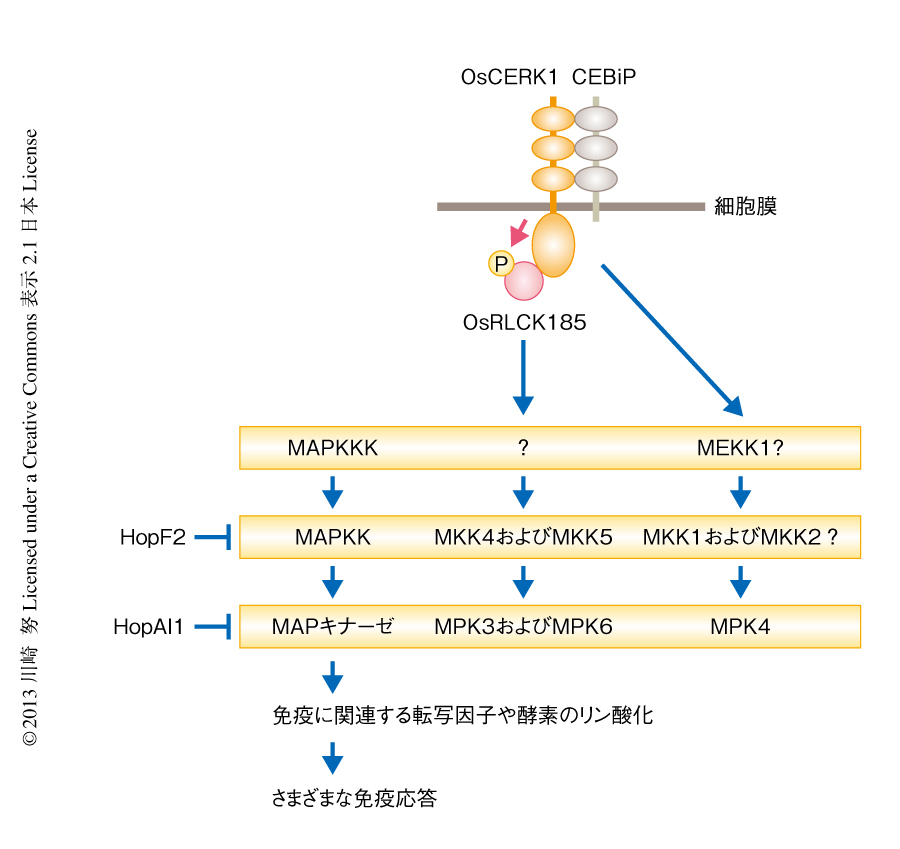
flg22やキチンなどのPAMPに応答してMAPキナーゼであるMPK3,MPK4,MPK6が活性化されることが知られている30)(図5).MPK4の上流にはMAPKKであるMKK1とMKK2,MAPKKKであるMEKK1が存在し,MEKK1-MKK1/2-MPK4というシグナル伝達経路の存在が示唆されている.一方,MPK3とMPK6の上流にはMAPKKであるMKK4とMKK5が位置していることが報告されているが,MAPKKKに関しては不明である.このようなMAPキナーゼカスケードの活性化はflg22やキチンを処理したのち10分以内と非常に早期に起こるため,パターン認識受容体からのシグナルは転写を介すことなくタンパク質間相互作用によりすぐにMAPキナーゼカスケードに伝達されていると考えられる.FLS2からのシグナルはBIK1,PBL1,PBL2,PBS1,BSK1などのRLCKファミリータンパク質を介して細胞に伝達されるため,それらRLCKファミリータンパク質がMAPキナーゼカスケードの活性化に関係していると考えられる.そのうち,BIK1,PBL1,BSK1のノックアウト変異体およびダブルノックアウト変異体について解析されたが,flg22に応答したMAPキナーゼの活性化に変化はみられず6,20),現在のところ,FLS2とMAPキナーゼカスケードとのあいだに存在するタンパク質はみつかっていない.
OsRLCK185はOsCERK1からのシグナルを細胞内に伝達しているため,筆者らは,OsRLCK185のノックダウン株を用いてキチンに応答したMAPキナーゼの活性化を解析した8)(新着論文レビュー でも掲載).その結果,OsRLCK185のノックダウン株ではキチンに応答したMPK3およびMPK6の活性化が抑制されていたが,MPK4の活性化は影響をうけないことがわかった.MPK3およびMPK6とMPK4は異なるカスケードにより活性化されると考えられたことから31),OsRLCK185はMPK3およびMPK6のカスケードの上流に位置しているが,MPK4のカスケードの上流には存在しないことがわかった.OsCERK1とMPK4のあいだには未同定のRLCKファミリータンパク質が存在していると思われた.いずれにしても,パターン認識受容体とMAPキナーゼカスケードを結ぶはじめてのタンパク質としてOsRLCK185が同定された.今後,OsRLCK185によるMAPキナーゼカスケードの活性化機構を解析することにより,これまで不明であったパターン認識受容体からMAPキナーゼカスケードの活性化にいたるシグナル伝達系を解明できるとものと期待している.
パターン認識受容体の下流ではMAPキナーゼカスケードが活性化され,それにともない転写因子の活性化や活性酸素の産生など,さまざまな免疫応答が誘導される.したがって,病原微生物にとってMAPキナーゼカスケードの活性化を抑制することは宿主の免疫応答を阻止するために非常に重要であると考えられる.P. syringaeのエフェクターHopAI1はリン酸化スレオニンリアーゼというまったく新しい酵素活性をもち,MAPキナーゼのもつ活性化ループにあるリン酸化スレオニン残基からリン酸基を非可逆的に取り除くことがわかった(図5).HopAI1はこの反応により活性化したMAPキナーゼをすぐに不活性化することで,MAPキナーゼを介したシグナル伝達系を阻害していると考えられる32).HopAI1と同じ酵素活性をもつエフェクターは動物に感染する赤痢菌からもみつかっており33),植物と動物において同じ活性によりMAPキナーゼの活性は抑制されていた.また,HopF2はMAPKKであるMKK5の機能発現のため必要なアルギニン残基をADPリボース化することにより,MAPキナーゼカスケードを阻害していることが報告されている34).
近年の研究により,植物および動物において病原微生物の認識にかかわる受容体が明らかにされ,病原微生物の感染にともなう免疫応答の誘導機構が少しずつ解明されてきた.なかでも,植物と動物が同じタンパク質ドメインから構成されるパターン認識受容体および細胞内受容体をもち,きわめて似たシステムにより病原微生物を認識し免疫応答を誘導していることは驚きである.まだ解析されている受容体はほんの一部にすぎないが,今後,さらに研究が進むにつれ,植物と動物における病原微生物の認識機構の相違点が明らかにされてくるであろう.
また,病原微生物が宿主の細胞内にエフェクターを送り込み免疫応答を抑制していることも明らかになってきた.ここでは,III型分泌機構により分泌されているエフェクターについて紹介したが,細菌,真菌,ウイルスなどさまざまな病原微生物が多様なエフェクターをもち,それらの機能はほとんど明らかになっていない.エフェクターの多くは既知のタンパク質との相同性がみられず,エフェクターの解析により新たなタンパク質の活性が見い出される可能性もあり,タンパク質科学への貢献も期待できる.また,エフェクター自体あるいはその分泌システムを抑制できれば,病原微生物の感染の拡大を阻止することにもつながることが期待できる.
作物の生産において病害による損失は非常に大きな問題である.植物免疫の研究により得られた成果を利用して,本来,植物自体がもつ免疫活性を増強し,病害を抑える技術が開発されることを期待している.
略歴:1996年 九州大学大学院農学研究科にて博士号取得,同年 奈良先端科学技術大学院大学バイオサイエンス研究科 助手,2000年 米国North Carolina大学Chapel Hill校 博士研究員,2002年 奈良先端科学技術大学院大学バイオサイエンス研究科 助教授を経て,2010年より近畿大学農学部 教授.
研究テーマ:植物免疫における病原微生物の認識とシグナル伝達の分子機構,病原微生物による感染戦略の分子機構.
抱負:植物において病原微生物の認識から抵抗性の発現にいたる過程ではたらくタンパク質を単離し,そのタンパク質間ネットワークを明らかにすることにより植物免疫の全貌の解明をめざす.また,研究により得られた植物免疫の基礎情報をもとに,植物自体がもつ免疫反応を増強させた“環境にやさしい耐病性植物”を開発したい.
研究室URL:http://kawasakirice.web.fc2.com/index.html
© 2013 川崎 努 Licensed under CC 表示 2.1 日本
(近畿大学農学部 バイオサイエンス学科植物分子遺伝学研究室)
email:川崎 努
領域融合レビュー, 2, e008 (2013) DOI: 10.7875/leading.author.2.e008
Tsutomu Kawasaki: Plant immunity and inhibitory mechanism of host immunity by pathogens.
要 約
植物は細胞膜に存在する病原微生物の認識にかかわる受容体により,侵入してきた病原微生物の構成成分を検出し,さまざまな免疫応答を誘導する.近年,病原微生物の認識にかかわる受容体の構造と機能が明らかにされるとともに,それら受容体と複合体を形成するタンパク質が同定され,病原微生物の認識にともなう免疫誘導の分子機構が明らかになってきた.一方,病原微生物は植物の免疫誘導を抑制するため,エフェクターとよばれる自らのタンパク質を植物の細胞内に分泌し,病原微生物の認識にかかわる受容体を介した免疫反応の誘導を阻害していることがわかってきた.ここでは,受容体を介した植物の免疫誘導の分子機構と,エフェクターを利用した病原微生物の感染戦略に関して,最新の知見を解説する.
はじめに
毎年,世界の作物生産の約15%は病害により失われている.一方で,世界的に爆発的な人口の増加が予測されており,国連食糧農業機関の発表によると,2050年には世界の人口は90億人をこえ,現在より食糧の60%の増産が必要であると試算されている.しかし,単位面積あたりの作物の生産量を大幅に向上させることは非常にむずかしく,病害による15%の損失をいかに抑えるかが今後の重要な課題のひとつになっている.
野菜や庭木などを育てていると,いろいろな病気が発生しその対策に悩まされる.植物を侵している病原微生物も,細菌,カビ,ウイルスなど,さまざまである.病気になった植物をみていると,あたかも植物が病原微生物から一方的な攻撃をうけているようにみえるが,本当にそうであろうか.地球上には多くの病原微生物が存在しているが,それらがすべての植物に感染できるわけではなく,個々の病原微生物は特定の植物にしか感染できない.つまり,植物は多くの病原微生物の感染を阻止しているのである.では,植物はどのようにして病原微生物の感染を阻止しているのだろうか.動物では一般に“免疫”として,病原微生物の感染にともない防御応答を誘導することが知られている.植物も動物の免疫と同じような方法で病原微生物に対し防御応答を誘導しているのだろうか.
動物の免疫応答は自然免疫と獲得免疫とに大別される.獲得免疫は古くからよく知られている抗原抗体反応にもとづく免疫反応であり,病原微生物の感染から抗体ができるまで数週間を要する.一方,自然免疫は病原微生物の感染にともない迅速に誘導される免疫応答である.自然免疫反応では,宿主(感染をうける側を宿主という)の細胞膜に存在する受容体が,病原微生物の構成成分(PAMP:pathogen-associated molecular pattern,病原微生物関連分子パターン)を検出することにより誘導される1)(図1).代表的なPAMPとして細菌のべん毛タンパク質やリポ多糖などが知られている.動物の細胞の表面にはこのようなPAMPを認識するセンサー(受容体)が存在し,それらは総称してパターン認識受容体(pattern recognition receptor:PRR)とよばれている.また,パターン認識受容体がPAMPを検出することにより誘導される免疫反応をPAMP誘導免疫(PAMP-triggered immunity:PTI)とよぶ.動物のパターン認識受容体の代表としてToll様受容体(Toll-like receptor:TLR)が知られており,Bruce A. BeutlerとJules A. HoffmannはToll様受容体の発見により2011年にノーベル生理学・医学賞を受賞している.ヒトには11個のToll様受容体が存在し,それらがさまざまな種類のPAMPを認識しそのシグナルを細胞内に伝達することにより,病原微生物の感染に対し迅速な免疫反応の誘導を可能にしている.
一方,植物には動物の獲得免疫のような抗原抗体反応にもとづく防御応答は存在しない.しかし,植物も動物と同様にパターン認識受容体をもち,PAMPを検出して防御応答を誘導することができる.その反応は動物の自然免疫に非常に似ていることから,近年,“植物免疫”とよばれている.わが国ではまだこの植物免疫という言葉にはなじみがないように思われるが,国際的には“plant immunity”という用語はすでに定着している.
病原微生物にとり,感染した細胞において自らのPAMPが宿主のパターン認識受容体に検出されてしまうと防御応答をうけることになり,増殖することができなくなる.しかし,病原微生物はただ手をこまねいているわけではない.病原微生物は進化により宿主の免疫応答を阻止することのできるタンパク質(エフェクターとよばれる)を獲得し,それを宿主の細胞内に送り込むことにより防御応答からまぬがれている1)(図1).それに対しさらに植物は,病原微生物が送りこんできたエフェクターを検知する細胞内受容体を獲得し,PAMP誘導免疫と比較してより強い免疫反応を誘導する.この反応はエフェクター誘導免疫(effector-triggered immunity:ETI)とよばれている(図1).では,このような植物の免疫反応と病原微生物による免疫反応の阻害はどのように行われているのだろうか.くわしくみてみよう.
1.植物のパターン認識受容体と免疫反応の誘導
植物の細胞膜に存在するパターン認識受容体は,受容体型キナーゼ(receptor-like kinase:RLK)と受容体型タンパク質(receptor-like protein:RLP)とに大きく分類される2)(図2).受容体型キナーゼはPAMPを検出する細胞外ドメインと細胞内にタンパク質キナーゼドメインをもつ受容体であり,一方,受容体型タンパク質は細胞外ドメインをもつものの細胞内の領域は短く特徴的なドメインをもたない.2000年以降,植物では細菌のべん毛タンパク質に由来するペプチド(flg22),翻訳伸長因子に由来するペプチド(elf18),細胞壁成分であるペプチドグリカン,そして,真菌のもつキチンを検出するパターン認識受容体が明らかになり先行して研究が進められているが,現在でも,パターン認識受容体とPAMPとの組合せが明らかになっているものはそれほど多くない.パターン認識受容体のもつ代表的な細胞外ドメインとしては,ロイシン残基のくり返し配列であるロイシンリッチリピート(leucine rich repeat:LRR),キチンやペプチドグリカンなどのオリゴ糖と結合するLysMドメインなどがある.植物のパターン認識受容体とヒトのToll様受容体の構造をみてみると,Toll様受容体のもつ細胞外ドメインはロイシンリッチリピートであり,植物のパターン認識受容体と非常によく似ていることがわかる(図2).
つぎに,このようなパターン認識受容体がどのようにPAMPを検出し,そのシグナルを細胞内に伝達するかをみてみよう.植物でもっともよく解析されているパターン認識受容体は,モデル植物であるシロイヌナズナ(Arabidopsis thaliana)のFLS2である.FLS2は細胞外にロイシンリッチリピートを,細胞内にタンパク質キナーゼドメインをもち,細菌のべん毛タンパク質のN末端領域に高度に保存されている22個のアミノ酸残基からなるペプチドflg22を検出する(図3).FLS2はflg22の非存在下では細胞膜においてホモ複合体を形成しているが3),flg22を検出するとやはり細胞外にロイシンリッチリピートをもつ受容体型キナーゼであるBAK1と相互作用する4).このFLS2とBAK1との相互作用は,細胞にflg22を処理したのち数分以内に起こることが報告されている.この相互作用によりFLS2とBAK1は相互にリン酸化され活性化状態になると考えられる.そののち,FLS2からのシグナルは受容体型細胞質タンパク質キナーゼ(RLCK:receptor-like cytoplasmic kinase)ファミリーに属するBIK1,PBS1,PBL1,PBL2,BSK1に伝達される5,6).これらRLCKファミリータンパク質はflg22の処理にともないリン酸化されることが明らかになっているが,FLS2とBAK1のどちらがRLCKをリン酸化しているかについては議論があり最終的な結論は得られていない.いずれにしても,これらのRLCKファミリータンパク質が受容体からのシグナルを受け取り,そのシグナルを細胞内に伝達していることはまちがいないと考えられる.ほかの植物にもFLS2やBAK1のホモログは存在するが,同様な機構によりflg22を認識しているのかどうかはわかっていない.また,ヒトのToll様受容体であるTLR5はべん毛タンパク質を検出するが,flg22とは異なるべん毛タンパク質の領域を検出することが明らかになっている.
真菌の成分であるキチンは代表的なPAMPとして植物に強い免疫応答を誘導する.イネにおけるキチンの検出にはパターン認識受容体であるOsCERK1とCEBiPが関与している(図3).OsCERK1は細胞外にLysMドメイン,細胞内にタンパク質キナーゼドメインをもち,CEBiPは細胞外にLysMドメインをもつものの,細胞内の領域は短く特異的なドメインはもたない7).CEBiPはキチンとの強い結合活性をもち,キチンを検出したCEBiPはOsCERK1と相互作用する.このことから,OsCERK1がキチンを検出したCEBiPからシグナルをうけ,そのシグナルを細胞内に伝達していると考えられていたが,その詳細は不明であった.筆者らは,OsCERK1の細胞内のタンパク質キナーゼドメインに結合するタンパク質として,RLCKファミリーに属するOsRLCK185を同定した8)(新着論文レビュー でも掲載).そして実際に,細胞膜においてOsCERK1とOsRLCK185とが相互作用していることを示した.さらに,OsRLCK185の発現をノックダウンしたイネの培養細胞ではキチンに応答した免疫応答が抑制され,OsRLCK185がOsCERK1からのシグナルを伝達する役割をはたしていることが明らかになった.
OsRLCK185はOsCERK1のタンパク質キナーゼドメインと結合していたことから,OsCERK1からOsRLCK185へのシグナル伝達はOsCERK1がOsRLCK185をリン酸化することにより行われている可能性が考えられた.そこで,キチンに応答したOsRLCK185のリン酸化について調べてみると,イネの培養細胞にキチンを処理したのち約5分でOsRLCK185はリン酸化されていた.さらに,OsCERK1およびOsRLCK185の組換えタンパク質を用いた解析により,OsCERK1がOsRLCK185を直接にリン酸化することが明らかになった.また一般に,RLCKファミリータンパク質は活性化ループとよばれる領域をもち,その活性化ループに存在するセリン残基とスレオニン残基のリン酸化が活性化に重要であると報告されている.そこで,OsRLCK185のもつ活性化ループのセリン残基とスレオニン残基をアラニン残基に置換したうえで,OsCERK1によるリン酸化実験を行ったところ,このOsRLCK185変異体はリン酸化されなかった.すなわち,OsCERK1はOsRLCK185の活性化ループに存在するセリン残基とスレオニン残基をリン酸化することによりOsRLCK185を活性化していると考えられた.
このように,キチンにより誘導される免疫応答では,キチンを受容したCEBiPがOsCERK1にシグナルを伝達し,OsCERK1はOsRLCK185を直接にリン酸化することにより細胞内にシグナルを伝達していることが明らかになった.このように,FLS2やOsCERK1などのパターン認識受容体からのシグナルはRLCKファミリータンパク質に伝達されていることから,ほかのパターン認識受容体においても同様にRLCKファミリータンパク質を介して細胞内にシグナルが伝達されている可能性が考えられる.イネには379個のRLCKファミリー遺伝子が存在し9),シロイヌナズナにも200個のRLCKファミリー遺伝子が存在しているが10),ほとんどのRLCKファミリータンパク質の機能は明らかになっておらず,今後の研究が期待される.
最近,OsCERK1から細胞内シグナル伝達には,低分子量Gタンパク質であるOsRac1を介したシグナル伝達も寄与していることが明らかになった11)(新着論文レビュー でも掲載).これまでの研究により,OsRac1はさまざまな免疫応答を誘導するシグナル伝達タンパク質と機能していることが示されており,OsCERK1は,OsRac1を活性化するGDP-GTP交換因子(GDP-GTP exchange factor:GEF)を直接にリン酸化することにより,OsRac1を介したシグナル伝達を活性化していることが明らかになった.
シロイヌナズナではOsCERK1のホモログであるAtCERK1がキチンを認識することが明らかになっている.イネのOsCERK1のもつLysMドメインはキチンとは結合しないためキチンに結合したCEBiPからシグナルを受け取るが,シロイヌナズナのAtCERK1のもつLysMドメインはキチンと強く結合し,ホモ複合体を形成することによりキチンを認識していることが明らかになった12).また,CEBiPと似た構造をもつシロイヌナズナのAtLYM1とAtLYM3は,PAMPのひとつである細菌の細胞壁成分ペプチドグリカンと結合し,そのシグナルをAtCERK1を介し細胞内に伝達していることが明らかになった13).イネにおいても,AtLYM1およびAtLYM3のホモログであるOsLYP4およびOsLYP6がペプチドグリカンと結合することが明らかになっており14),シロイヌナズナと同様に,OsLYP4とOsLYP6がペプチドグリカンを検出し,そのシグナルをOsCERK1を介して細胞内に伝達していると推測されているが,実際に,OsCERK1がペプチドグリカンの認識にかかわるかどうかは証明されていない.しかし,OsRLCK185のノックダウン株ではペプチドグリカンに応答した免疫応答が抑制されており,OsCERK1がペプチドグリカンの認識に関与することが強く示唆されている.このように,CERK1は真菌のPAMPであるキチンおよび細菌のPAMPであるペプチドグリカンを検出するパターン認識受容体であり,真菌と細菌の両方に対し免疫応答を誘導していると考えられる.
筆者らは,OsRLCK185のシロイヌナズナにおけるオーソログとしてAtRLCK1を同定している.AtRLCK1は,イネのOsRLCK185と同様にCERK1に結合し,AtRLCK1の機能欠損変異体ではキチンに応答した免疫応答は阻害されていることがわかった(未発表).このように,イネとシロイヌナズナにはキチンに対する応答に関与する共通のシグナル伝達系が存在していると思われる.
2.植物の免疫応答を阻止するエフェクター
植物はパターン認識受容体により病原微生物の感染を認識し,さまざまな免疫応答を誘導して病原微生物の増殖を阻止している.では,病原微生物はこの植物の免疫応答にどのように対抗しているのだろうか.病原微生物は宿主への感染過程において自らのタンパク質を宿主の細胞内に分泌し免疫応答を抑制している(図1).このような分泌タンパク質は総称してエフェクターとよばれている.病原微生物の種類によりエフェクターの分泌の様式は異なるが,ここでは,一部の病原細菌が利用するIII型分泌装置について紹介したい.Pseudomonas属やXanthomonas属の病原細菌は針状の構造をしたIII型分泌装置を用いて宿主の細胞内にエフェクターを分泌している.III型分泌装置を構成するタンパク質をコードする遺伝子はhrp(hypersensitive response and pathogenicity)遺伝子とよばれ,このhrp遺伝子は植物へ感染するときのみ発現する.つまり,病原細菌は宿主の組織内の環境を感知してIII型分泌装置を構築し,宿主の細胞内にエフェクターを送り込んでいるのである.
Pseudomonas属やXanthomonas属の細菌は30~50個のエフェクターをもち,III型分泌装置を使いこれを宿主の細胞内に送り込んでいる.しかし,アミノ酸配列から機能を推定できるエフェクターは少なく,作用機作のわかっているエフェクターの数も少ない.また,エフェクターの多くは既知の機能ドメインをもたないことから,進化的に新しいタンパク質であると考えられている.実際,近年になり,エフェクターの研究から新規な酵素活性が見い出されている.また,多くのエフェクターは宿主の細胞内で発現させるとその免疫応答を強く阻害する.このことは,宿主において免疫応答の誘導に重要な因子の機能をエフェクターが阻害していることを示唆している.そのため近年,エフェクターが標的とする因子を探索することにより,免疫応答において重要な役割をはたしている因子を同定しようという試みが行われている.その具体例について紹介していこう.
斑葉細菌病をひき起こすPseudomonas syringaeはグラム陰性細菌であり,古くからシロイヌナズナを用いた免疫の研究において代表的な病原微生物として利用されている.P. syringaeのエフェクターHopM1の標的となる因子として,シロイヌナズナのMIN7遺伝子がクローニングされた15).MIN7遺伝子はARFのGDP-GTP交換因子をコードしており,PAMP誘導性免疫やエフェクター誘導免疫に関与する小胞輸送を制御することが示唆されている16).また,P. syringaeのエフェクターHopU1の標的となる因子として,シロイヌナズナにおいてRNA結合タンパク質をコードするGRP7遺伝子がクローニングされた17).HopU1はGRP7のRNA認識モチーフに存在するアルギニン残基をADPリボース化することにより,GRP7のRNA結合活性を阻害していることが報告された18).さらに最近,GRP7はパターン認識受容体であるFLS2の転写産物や翻訳関連タンパク質と相互作用することにより転写後制御に関与しており19),HopU1はGRP7とFLS2の転写産物とのあいだの相互作用を阻害することにより,FLS2のタンパク質量を減少させていることが報告された.
FLS2やCERK1による免疫誘導機構の解析から明らかになったように,RLCKファミリータンパク質はパターン認識受容体からのシグナルを細胞内に伝達する重要なタンパク質である.近年,エフェクターが相互作用するタンパク質の探索により,RLCKファミリータンパク質がさまざまなエフェクターの標的となることも明らかになってきた.P. syringaeのエフェクターAvrPphBはシステインプロテアーゼ活性をもち,免疫応答に関与するRLCKファミリータンパク質であるBIK1やPBS1,PBL1,PBL2を切断することにより免疫応答を抑制していることが明らかになった5)(図4).また,Xanthomonas campestrisのエフェクターであるAvrACは,RLCKファミリーに属するRIPKおよびBIK1を標的としていることが明らかになった20).RLCKファミリータンパク質の活性化においてその活性化ループに存在するセリン残基やスレオニン残基のリン酸化が重要であると考えられるが,AvrACはそのリン酸化部位にウリジル酸を付加することによりRIPKおよびBIK1の活性化を阻害していた.また,RLCKファミリーのほかのタンパク質においても活性化ループのリン酸化部位は高度に保存されており,それらがAvrACの標的となる可能性も考えられる.また,筆者らは,イネ白葉枯病菌Xanthomonas oryzaeのエフェクターであるXoo1488がOsRLCK185を標的としていることを明らかにした.OsCERK1はOsRLCK185をリン酸化することによりシグナルを伝達しているが,Xoo1488はOsCERK1によるOsRLCK185のリン酸化を阻害していた8)(新着論文レビュー でも掲載).このように,複数のエフェクターがRLCKファミリータンパク質を標的としていることが明らかになり,このことは,RLCKファミリータンパク質が免疫応答において重要な役割をはたしていることを裏づけるものと考えられる.
P. syringaeのエフェクターHopZ1は抗菌性物質であるダイドゼインの合成に関与する2-ヒドロキシイソフラバノンデヒドラターゼを標的としていることが明らかになった21).免疫応答の際に産生される抗菌性物質はファイトアレキシンとよばれ,その産生は免疫応答における重要な反応のひとつである.HopZ1はファイトアレキシンの産生を抑制することにより免疫応答を阻害していると考えられる21).
また,エフェクターがパターン認識受容体を直接に抑制することも報告されている(図4).P. syringaeのエフェクターであるAvrPtoおよびAvrPtoBは,FLS2やEFR,CERK1と直接に相互作用し免疫誘導を阻害していることが示された22-25).さらに,AvrPtoBはBAK1も阻害していることが明らかになった26).BAK1は複数のパターン認識受容体と協調して機能していることから,病原微生物にとりBAK1の機能を抑制することにより複数のパターン認識受容体を阻害することが可能になり,免疫応答を効率的に抑制することができるものと考えられる.
Xanthomonas属の細菌はTAL(transcription activator-like)とよばれるエフェクターをもつ.このTALエフェクターは宿主のもつDNA配列を特異的に認識する反復配列と,核移行シグナルおよび転写活性化ドメインをもつ転写因子である.反復配列は34アミノ酸残基からなり,その12番目と13番目のアミノ酸残基が認識する塩基配列の特異性を決めている.最近,X. oryzaeのTALエフェクターであるAvrXa7は,SWEETSとよばれる糖輸送体Os8N3およびOs11N3の遺伝子の転写を活性化することが報告された27).AvrXa7は自らの増殖に必要な養分を得るため,宿主のもつ糖輸送体を活性化していると考えられる.また,TALエフェクターのDNA結合領域を利用すると,任意の塩基配列に結合するDNA結合タンパク質を構築することができる.これを使い人工酵素であるTALエフェクターヌクレアーゼ(transcription activator-like effector nuclease:TALEN)を作製し,それを細胞に導入することにより目的の遺伝子を改変した生物が作出できるようになった.このように,TALエフェクターは宿主において転写を制御することにより免疫応答の抑制,あるいは,自らの増殖に適した環境をつくりだしている.
以上のように,個々のエフェクターの機能が少しずつ明らかにされ,さらに,エフェクターを分子プローブとした新規な因子の探索により,これまで明らかにされていなかった免疫にかかわる因子が同定されている.しかし,解析されたエフェクターはまだ一部にすぎず,いまだ,多くのエフェクターの機能,あるいは,宿主における標的は不明である.宿主と病原微生物との攻防を分子レベルで解明するためには,さらなる研究が必要である.
3.細胞内受容体と免疫誘導
植物および動物の細胞膜にはパターン認識受容体が存在しPAMPを検出して防御応答を誘導していることを説明してきたが,植物および動物は細胞内にも共通の構造をもつ細胞内受容体をもつ(図2).この細胞内受容体は中心部に核酸結合ドメイン,C末端側にロイシンリッチリピートをもつ.この核酸結合ドメインは,植物ではNBドメイン(NB:nucleotide-binding),動物ではNOD(nucleotide-binding oligomerization domain,ヌクレオチド結合オリゴマー形成ドメイン)とよばれている1).植物の細胞内受容体の多くはN末端側にコイルドコイルドメインあるいはTIRドメインをもつ.一方,動物の細胞内受容体のN末端側にはカスパーゼ結合ドメイン(caspase recruitment domain:CARD)などが存在する.これらN末端側の領域はタンパク質間相互作用に関係し,下流にシグナルを伝達するためのドメインとして機能していると考えられており,動物ではCARDを介した免疫応答の活性化機構が明らかにされている.しかし,植物では細胞内受容体から下流にどのようにシグナルが伝達されているか,ブラックボックスのままである.
植物の細胞内受容体はもともと,病原微生物の感染にともない強い抵抗性反応を誘導する抵抗性遺伝子座にコードされるタンパク質(抵抗性タンパク質,あるいは,Rタンパク質ともよばれる)として同定された.のちの研究により,抵抗性タンパク質として機能する細胞内受容体は病原微生物のエフェクターを特異的に認識し,過敏感細胞死をともなう強い免疫反応(エフェクター誘導免疫)を誘導することが明らかになった(図1).植物の細胞内受容体の認識においては,細胞内受容体とエフェクターとが直接的に相互作用する場合と,エフェクターと宿主のほかのタンパク質との相互作用を細胞内受容体が検出することによりエフェクターを間接的に認識する場合とがある.たとえば,シロイヌナズナの細胞内受容体であるRPS5はP. syringaeのエフェクターAvrPphBがRLCKファミリータンパク質であるPBS1を切断するのを検出して免疫反応を誘導する28).また,P. syringaeのエフェクターAvrBはRLCKファミリータンパク質であるRIPKによるRIN4のリン酸化を促進し,細胞内受容体であるRPM1はリン酸化されたRIN4を検出することにより免疫応答を誘導している29).このように,植物の細胞内受容体はエフェクターを検出して免疫を誘導しているが,動物の細胞内受容体ではエフェクターではなくPAMPを検出して免疫応答を誘導することが知られている.今後,研究が進むにつれ,植物の細胞内受容体によるPAMPの認識,あるいは,動物の細胞内受容体によるエフェクターの認識が明らかになるかもしれない.
4.MAPキナーゼを介した免疫誘導機構
MAPキナーゼは酵母から植物あるいは動物にいたるまで真核生物に広く保存されたセリン/スレオニンキナーゼである.MAPキナーゼは免疫応答を含むさまざまな生理反応において活性化され,活性化されたMAPキナーゼは転写因子や酵素などをリン酸化することにより細胞内シグナル伝達を担うモジュールとして機能している.MAPキナーゼはMAPキナーゼキナーゼ(MAPKK)がキナーゼサブドメインVIIとキナーゼサブドメインVIIIとのあいだにあるTxYモチーフのスレオニン残基およびチロシン残基をリン酸化することにより活性化される.さらに,MAPKKはMAPキナーゼキナーゼキナーゼ(MAPKKK)がキナーゼサブドメインVIIとキナーゼサブドメインVIIIとのあいだにあるSxxxS/Tモチーフのセリン残基およびスレオニン残基をリン酸化することにより活性化される30).これら3種類のタンパク質キナーゼのリン酸化によるシグナル伝達はMAPキナーゼカスケードとよばれている(図5).シロイヌナズナには60個のMAPKKK,10個のMAPKK,20個のMAPキナーゼが,イネには75個のMAPKKK,8個のMAPKK,15個のMAPキナーゼが同定されており,その数の多さから複雑なカスケードが想像できるが,どのようにシグナルが伝達されているかについて,その詳細は不明である.
flg22やキチンなどのPAMPに応答してMAPキナーゼであるMPK3,MPK4,MPK6が活性化されることが知られている30)(図5).MPK4の上流にはMAPKKであるMKK1とMKK2,MAPKKKであるMEKK1が存在し,MEKK1-MKK1/2-MPK4というシグナル伝達経路の存在が示唆されている.一方,MPK3とMPK6の上流にはMAPKKであるMKK4とMKK5が位置していることが報告されているが,MAPKKKに関しては不明である.このようなMAPキナーゼカスケードの活性化はflg22やキチンを処理したのち10分以内と非常に早期に起こるため,パターン認識受容体からのシグナルは転写を介すことなくタンパク質間相互作用によりすぐにMAPキナーゼカスケードに伝達されていると考えられる.FLS2からのシグナルはBIK1,PBL1,PBL2,PBS1,BSK1などのRLCKファミリータンパク質を介して細胞に伝達されるため,それらRLCKファミリータンパク質がMAPキナーゼカスケードの活性化に関係していると考えられる.そのうち,BIK1,PBL1,BSK1のノックアウト変異体およびダブルノックアウト変異体について解析されたが,flg22に応答したMAPキナーゼの活性化に変化はみられず6,20),現在のところ,FLS2とMAPキナーゼカスケードとのあいだに存在するタンパク質はみつかっていない.
OsRLCK185はOsCERK1からのシグナルを細胞内に伝達しているため,筆者らは,OsRLCK185のノックダウン株を用いてキチンに応答したMAPキナーゼの活性化を解析した8)(新着論文レビュー でも掲載).その結果,OsRLCK185のノックダウン株ではキチンに応答したMPK3およびMPK6の活性化が抑制されていたが,MPK4の活性化は影響をうけないことがわかった.MPK3およびMPK6とMPK4は異なるカスケードにより活性化されると考えられたことから31),OsRLCK185はMPK3およびMPK6のカスケードの上流に位置しているが,MPK4のカスケードの上流には存在しないことがわかった.OsCERK1とMPK4のあいだには未同定のRLCKファミリータンパク質が存在していると思われた.いずれにしても,パターン認識受容体とMAPキナーゼカスケードを結ぶはじめてのタンパク質としてOsRLCK185が同定された.今後,OsRLCK185によるMAPキナーゼカスケードの活性化機構を解析することにより,これまで不明であったパターン認識受容体からMAPキナーゼカスケードの活性化にいたるシグナル伝達系を解明できるとものと期待している.
パターン認識受容体の下流ではMAPキナーゼカスケードが活性化され,それにともない転写因子の活性化や活性酸素の産生など,さまざまな免疫応答が誘導される.したがって,病原微生物にとってMAPキナーゼカスケードの活性化を抑制することは宿主の免疫応答を阻止するために非常に重要であると考えられる.P. syringaeのエフェクターHopAI1はリン酸化スレオニンリアーゼというまったく新しい酵素活性をもち,MAPキナーゼのもつ活性化ループにあるリン酸化スレオニン残基からリン酸基を非可逆的に取り除くことがわかった(図5).HopAI1はこの反応により活性化したMAPキナーゼをすぐに不活性化することで,MAPキナーゼを介したシグナル伝達系を阻害していると考えられる32).HopAI1と同じ酵素活性をもつエフェクターは動物に感染する赤痢菌からもみつかっており33),植物と動物において同じ活性によりMAPキナーゼの活性は抑制されていた.また,HopF2はMAPKKであるMKK5の機能発現のため必要なアルギニン残基をADPリボース化することにより,MAPキナーゼカスケードを阻害していることが報告されている34).
おわりに
近年の研究により,植物および動物において病原微生物の認識にかかわる受容体が明らかにされ,病原微生物の感染にともなう免疫応答の誘導機構が少しずつ解明されてきた.なかでも,植物と動物が同じタンパク質ドメインから構成されるパターン認識受容体および細胞内受容体をもち,きわめて似たシステムにより病原微生物を認識し免疫応答を誘導していることは驚きである.まだ解析されている受容体はほんの一部にすぎないが,今後,さらに研究が進むにつれ,植物と動物における病原微生物の認識機構の相違点が明らかにされてくるであろう.
また,病原微生物が宿主の細胞内にエフェクターを送り込み免疫応答を抑制していることも明らかになってきた.ここでは,III型分泌機構により分泌されているエフェクターについて紹介したが,細菌,真菌,ウイルスなどさまざまな病原微生物が多様なエフェクターをもち,それらの機能はほとんど明らかになっていない.エフェクターの多くは既知のタンパク質との相同性がみられず,エフェクターの解析により新たなタンパク質の活性が見い出される可能性もあり,タンパク質科学への貢献も期待できる.また,エフェクター自体あるいはその分泌システムを抑制できれば,病原微生物の感染の拡大を阻止することにもつながることが期待できる.
作物の生産において病害による損失は非常に大きな問題である.植物免疫の研究により得られた成果を利用して,本来,植物自体がもつ免疫活性を増強し,病害を抑える技術が開発されることを期待している.
文 献
- Jones, J. D. & Dangl, J. L.: The plant immune system. Nature, 444, 323-329 (2006)[PubMed]
- Monaghan, J. & Zipfel, C.: Plant pattern recognition receptor complexes at the plasma membrane. Curr. Opin. Plant Biol., 15, 349-357 (2012)[PubMed]
- Sun, W., Cao, Y., Jansen Labby, K. et al.: Probing the Arabidopsis flagellin receptor: FLS2-FLS2 association and the contributions of specific domains to signaling function. Plant Cell, 24, 1096-1113 (2012)[PubMed]
- Chinchilla, D., Zipfel, C., Robatzek, S. et al.: A flagellin-induced complex of the receptor FLS2 and BAK1 initiates plant defence. Nature, 448, 497-500 (2007)[PubMed]
- Zhang, J., Li, W., Xiang, T. et al.: Receptor-like cytoplasmic kinases integrate signaling from multiple plant immune receptors and are targeted by a Pseudomonas syringae effector. Cell Host Microbe, 7, 290-301 (2010)[PubMed]
- Shi, H., Shen, Q., Qi, Y. et al.: BR-signaling kinase1 physically associates with Flagellin Sensing2 and regulates plant innate immunity in Arabidopsis. Plant Cell, 25, 1143-1157 (2013)[PubMed]
- Shimizu, T., Nakano, T., Takamizawa, D. et al.: Two LysM receptor molecules, CEBiP and OsCERK1, cooperatively regulate chitin elicitor signaling in rice. Plant J., 64, 204-214 (2010)[PubMed]
- Yamaguchi, K., Yamada, K., Ishikawa, K. et al.: A receptor-like cytoplasmic kinase targeted by a plant pathogen effector is directly phosphorylated by the chitin receptor and mediates rice immunity. Cell Host Microbe, 13, 347-357 (2013)[PubMed] [新着論文レビュー]
- Vij, S., Giri, J., Dansana, P. K. et al.: The receptor-like cytoplasmic kinase (OsRLCK) gene family in rice: organization, phylogenetic relationship, and expression during development and stress. Mol. Plant, 1, 732-750 (2008)[PubMed]
- Jurca, M. E., Bottka, S. & Feher, A.: Characterization of a family of Arabidopsis receptor-like cytoplasmic kinases (RLCK class VI). Plant Cell Rep., 27, 739-748 (2008)[PubMed]
- Akamatsu, A., Wong, H. L., Fujiwara, M. et al.: An OsCEBiP/OsCERK1-OsRacGEF1-OsRac1 module is an essential early component of chitin-induced rice immunity. Cell Host Microbe, 13, 465-476 (2013)[PubMed] [新着論文レビュー]
- Liu, T., Liu, Z., Song, C. et al.: Chitin-induced dimerization activates a plant immune receptor. Science, 336, 1160-1164 (2012)[PubMed]
- Willmann, R., Lajunen, H. M., Erbs, G. et al.: Arabidopsis lysin-motif proteins LYM1 LYM3 CERK1 mediate bacterial peptidoglycan sensing and immunity to bacterial infection. Proc. Natl. Acad. Sci. USA, 108, 19824-19829 (2011)[PubMed]
- Liu, B., Li, J. F., Ao, Y. et al.: Lysin motif-containing proteins LYP4 and LYP6 play dual roles in peptidoglycan and chitin perception in rice innate immunity. Plant Cell, 24, 3406-3419 (2012)[PubMed]
- Nomura, K., Debroy, S., Lee, Y. H. et al.: A bacterial virulence protein suppresses host innate immunity to cause plant disease. Science, 313, 220-223 (2006)[PubMed]
- Nomura, K., Melotto, M., He, S. Y.: Suppression of host defense in compatible plant-Pseudomonas syringae interactions. Curr. Opin. Plant Biol., 8, 361-368 (2005)[PubMed]
- Fu, Z. Q., Guo, M., Jeong, B. R. et al.: A type III effector ADP-ribosylates RNA-binding proteins and quells plant immunity. Nature, 447, 284-288 (2007)[PubMed]
- Jeong, B. R., Lin, Y., Joe, A. et al.: Structure function analysis of an ADP-ribosyltransferase type III effector and its RNA-binding target in plant immunity. J. Biol. Chem., 286, 43272-43281 (2011)[PubMed]
- Nicaise, V., Joe, A., Jeong, B. R. et al.: Pseudomonas HopU1 modulates plant immune receptor levels by blocking the interaction of their mRNAs with GRP7. EMBO J., 32, 701-712 (2013)[PubMed]
- Feng, F., Yang, F., Rong, W. et al.: A Xanthomonas uridine 5'-monophosphate transferase inhibits plant immune kinases. Nature, 485, 114-118 (2012)[PubMed]
- Zhou, H., Lin, J., Johnson, A. et al.: Pseudomonas syringae type III effector HopZ1 targets a host enzyme to suppress isoflavone biosynthesis and promote infection in soybean. Cell Host Microbe, 9, 177-186 (2011)[PubMed]
- Xiang, T., Zong, N., Zou, Y. et al.: Pseudomonas syringae effector AvrPto blocks innate immunity by targeting receptor kinases. Curr. Biol., 18, 74-80 (2008)[PubMed]
- Xiang, T., Zong, N., Zhang, J. et al.: BAK1 is not a target of the Pseudomonas syringae effector AvrPto. Mol Plant Microbe Interact., 24, 100-107 (2011)[PubMed]
- Gohre, V., Spallek, T., Haweker, H. et al.: Plant pattern-recognition receptor FLS2 is directed for degradation by the bacterial ubiquitin ligase AvrPtoB. Curr. Biol., 18, 1824-1832 (2008)[PubMed]
- Gimenez-Ibanez, S., Hann, D. R., Ntoukakis, V. et al.: AvrPtoB targets the LysM receptor kinase CERK1 to promote bacterial virulence on plants. Curr. Biol., 19, 423-429 (2009)[PubMed]
- Shan, L., He, P., Li, J. et al.: Bacterial effectors target the common signaling partner BAK1 to disrupt multiple MAMP receptor-signaling complexes and impede plant immunity. Cell Host Microbe, 4, 17-27 (2008)[PubMed]
- Chen, L. Q., Hou, B. H., Lalonde, S. et al.: Sugar transporters for intercellular exchange and nutrition of pathogens. Nature, 468, 527-532 (2010)[PubMed]
- Shao, F., Golstein, C., Ade, J. et al.: Cleavage of Arabidopsis PBS1 by a bacterial type III effector. Science, 301, 1230-1233 (2003)[PubMed]
- Liu, J., Elmore, J. M., Lin, Z. J. et al.: A receptor-like cytoplasmic kinase phosphorylates the host target RIN4, leading to the activation of a plant innate immune receptor. Cell Host Microbe, 9, 137-146 (2011)[PubMed]
- Tena, G., Boudsocq, M. & Sheen, J.: Protein kinase signaling networks in plant innate immunity. Curr. Opin. Plant Biol., 14, 519-529 (2011)[PubMed]
- Kishi-Kaboshi, M., Okada, K., Kurimoto, L. et al.: A rice fungal MAMP-responsive MAPK cascade regulates metabolic flow to antimicrobial metabolite synthesis. Plant J., 63, 599-612 (2010)[PubMed]
- Zhang, J., Shao, F., Li, Y. et al.: A Pseudomonas syringae effector inactivates MAPKs to suppress PAMP-induced immunity in plants. Cell Host Microbe, 1, 175-185 (2007)[PubMed]
- Li, H., Xu, H., Zhou, Y. et al.: The phosphothreonine lyase activity of a bacterial type III effector family. Science, 315, 1000-1003 (2007)[PubMed]
- Wang, Y., Li, J., Hou, S. et al.: A Pseudomonas syringae ADP-ribosyltransferase inhibits Arabidopsis mitogen-activated protein kinase kinases. Plant Cell, 22, 2033-2044 (2010)[PubMed]
著者プロフィール
略歴:1996年 九州大学大学院農学研究科にて博士号取得,同年 奈良先端科学技術大学院大学バイオサイエンス研究科 助手,2000年 米国North Carolina大学Chapel Hill校 博士研究員,2002年 奈良先端科学技術大学院大学バイオサイエンス研究科 助教授を経て,2010年より近畿大学農学部 教授.
研究テーマ:植物免疫における病原微生物の認識とシグナル伝達の分子機構,病原微生物による感染戦略の分子機構.
抱負:植物において病原微生物の認識から抵抗性の発現にいたる過程ではたらくタンパク質を単離し,そのタンパク質間ネットワークを明らかにすることにより植物免疫の全貌の解明をめざす.また,研究により得られた植物免疫の基礎情報をもとに,植物自体がもつ免疫反応を増強させた“環境にやさしい耐病性植物”を開発したい.
研究室URL:http://kawasakirice.web.fc2.com/index.html
© 2013 川崎 努 Licensed under CC 表示 2.1 日本